Las oportunidades que ofrece la genómica y los retos que plantea en la mejora productiva del ovino lechero: mitos y realidades

SEMINARIO INTERNACIONAL PONENCIA INAUGURAL:
‘La genómica como herramienta para el desarrollo del ovino lechero en el mundo’
Juan José Arranz. Profesor Titular del Dpto de Producción Animal de la Universidad de León
Vamos a centrarnos en esta ponencia en aspectos básicos de la genómica, que quizás no estén del todo claros, de aplicaciones en el ovino lechero y también de mitos y realidades, es decir, de cuestiones prácticas como la selección genómica.
LA GENÓMICA COMO CIENCIA
La genómica es una ciencia que nace a finales del siglo XX, cuando se plantea el análisis y el estudio del genoma de un organismo. Por lo tanto, es la ciencia que estudia la información genética de cualquier especie de manera global, en su conjunto, tanto la estructura, la función, la evolución... Nace en el momento que el desarrollo tecnológico permite pasar de un estudio puntual, un gen o un fenotipo de manera individual, a un estudio general.
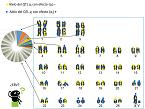
Sabemos que las células tienen un conjunto de información. En el caso de la oveja, esta información se estructura en 27 pares cromosómicos, un par que llamamos ‘sexuales’, pero que también tienen información, y el resto. La genómica pretende conocer la estructura, es decir, la secuencia de nucleóticos que hay en esos cromosomas, y la función de cada una de las partes. El genoma tiene una base común a la especie, pero también una diversidad, que es la base de las diferencias productivas o raciales.
EL GENOMA OVINO
La genómica en el ganado ovino es fruto, fundamentalmente, de un proceso de abaratamiento de la tecnología genómica. En perspectiva, el desarrollo del genoma humano es un proyecto que cuesta unos 3.000 millones de dólares. En vacuno, cuesta unos 15 o 20 millones de dólares en su etapa de desarrollo. Por su parte, el genoma de la oveja ha sido algo mucho más económico, por la posibilidad de aprovechar el desarrollo tecnológico para estudiar unas especies que, en principio, tienen menos importancia desde el punto de vista productivo.
Hoy en día, tenemos un borrador del genoma de la oveja bastante avanzado y una base de datos en la que se puede observar que hay más de 400 animales distintos secuenciados de varias razas, lo que representa bien la diversidad genética que tiene esta especie en nuestro planeta. Al analizar este conjunto de genomas, se observa que hay una gran parte que es común y otra parte, o una serie de puntos, que son variables. Así, existe una amplia variedad de poliformismos de un único nucléotido, o los llamados SNP, que son las siglas de ‘Single Nucleotide Polymorphism’, distribuidos entre los 27 cromosomas en los que está dividido el genoma de la oveja.
HERRAMIENTAS GENÓMICAS EN EL GANADO OVINO
A partir del conocimiento del genoma de la oveja, se desarrollan una serie de herramientas en estos conocimientos genómicos.
Uno de los más importantes son los chips de SNP, porque siguen una tecnología de producción similar a los microchips de los computadores, básicamente lo que tienen es un conjunto de marcadores que están distribuidos por todos los cromosomas. Como se tienen más de 50 millones de variantes, por lo que en función de la localización, se intenta balizar el genoma, poniendo unos puntos de referencia, de una manera más o menos equidistante que cubran las diferentes regiones de los cromosomas. De los 50 millones, se eligen un número, que pueden ser 50.000, se pone en un sistema sólido como el chip. En cada una de las doce celdillas del chip, se pone una sonda que se corresponde con las dos secuencias del SNP, por ejemplo, con el nucleótido A y el nucleótido G. Es una herramienta sólida que se elige porque están presentes en la mayoría de las razas que hemos seleccionado.
ELIMINACIÓN DE GENES LETALES EN EL GANADO OVINO
Existen una serie de aplicaciones en ganado ovino que permiten utilizar la variabilidad que se determina con el chip con determinados fines.
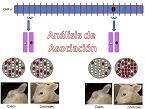
La primera que se hizo fue muy exitosa, ya que se trataba de detectar alelos letales que eran capaces de causar enfermedades hereditarias.
En la detección de enfermedades, se tiene un fenotipo muy claro, que son animales que están sanos y otros que están enfermos. Por lo tanto, se plantea la búsqueda de ese gen causante de esas enfermedades hereditarias y saber cuál es su mutación. Se divide el cromosoma en segmentos en un conjunto de individuos sanos y en otros que están enfermos. Se trata de un estudio que no es demasiado complejo, haciendo un análisis de asociación y se observa que en los SNP que no están asociados, la frecuencia o la distribución es más o menos en función de que los animales sean enfermos o sanos. Sin embargo, en aquellos que están en la proximidad del gen causal identificado, se tiene una clara distorsión en los alelos y está enriquecido uno de los alelos.
Con el gen y la mutación, se pueden eliminar de la reproducción los animales portadores sanos.
Es una aplicación sencilla y muy eficiente.
SELECCIÓN GENÓMICA
La selección genómica se puede definir como un proceso de estimación del valor genético de un individuo a partir de la información de un gran número de marcadores genéticos, los denominados SNP que aparecen en el chip. Así, se identifican los genes que controlan un determinado carácter. Se intenta hacer una asociación similar a la que se hace en las enfermedades hereditarias, para determinar que los que tienen un determinado nucleótido son más productores que otro. Hay diferencias, ya que en el caso de que haya un único gen el planteamiento es muy sencillo y la identificación suele ser exitosa. En este caso, no se plantea identificar los genes, sino dar un efecto debido a la proximidad del gen causal del marcador. Una vez que se tengan los efectos en una población denominada ‘de referencia’, se puede predecir en individuos relacionados con esta población su valor genético.
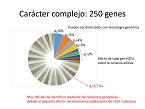
En el caso de la selección genómica, no se tiene un fenotipo fácil de identificar. Un animal no está sano o enfermo, o es de alta producción o de baja. Si se analiza la población, la mayoría de los individuos están alrededor de la media y hay pocos individuos que se encuentren muy por encima de la media y pocos individuos que prácticamente no produzcan. Desde el punto de vista genético, se debe a la actividad de un número elevado de genes, como efecto del ambiente en el que el animal está produciendo, a una serie de interacciones que se producen entre los genes... y determina un fenotipo complejo. Genéticamente, en lugar de un único punto, tenemos muchos genes QTL con alelos positivos y negativos. Se trata de seleccionar a aquellos genes que tienen una mayor proporción de alelos positivos.
La cuestión es compleja porque en el caso de las enfermedades hereditarias se tenía un gen que marcaba dos clases, pero en este caso hay 250 genes, con unos pocos que tienen un pequeño efecto infinitesimal de ese modelo. Por lo tanto, al total de la varianza genética, contribuyen muy poco. Si se hiciera un trabajo de asociación como hemos visto en las enfermedades hereditarias, quizás se podría identificar algunas de las regiones en las que están los genes, pero la mayoría son muy difíciles de identificar. Si se plantea a nivel teórico, tendríamos que analizar conjuntos de más de 25.000 o 100.000 animales. Identificar estos genes es una labor prácticamente imposible con unos medios económicos limitados y probablemente tampoco tenga un mayor interés.
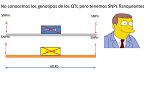
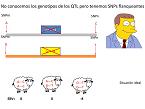
El valor genético de un animal sería hacer una suma de cada uno de los alelos del QTL para obtener ese valor genético teórico. Sin embargo, a nivel práctico es mucho más complicado, porque no se van a conocer dónde están ni qué efecto tienen la mayor parte de los genes. A nivel individual, si funcionara como un gen mayor, el fenotipo sería su valor aditivo más un porcentaje de error. Si se analiza en cuestión de genes, el efecto sería el valor aditivo de cada uno de los QTL. Sin embargo, nos encontramos con el problema de que es prácticamente imposible detectar todos los genes. A este problema se le da una solución, utilizando un número muy elevado de marcadores que flanquean a esos genes. A partir de ahí, se divide el genoma entero en segmentos y esos segmentos están flanqueados por los SNP que están en un chip y se va a evaluar con una población en la que tenemos medidas muy precisas de esos caracteres. Se conoce un genotipo y, por lo tanto, se da un efecto a cada uno de esos SNP a cada carácter. Es la solución que se propone a la falta de conocimiento de los QTL. Así, utilizamos un chip, dividimos cada genoma en segmentos e intentar dar a cada chip un efecto y estimar su valor genético. En cada segmento, se tiene un SNP. Se asume que en ese segmento puede haber un QTL que afecta al carácter cuantitativo y la distancia que se propone suele ser bastante pequeña, en concreto de unos 60.000 pares de bases.
No se conocen exactamente los QTL, sino que lo que se intenta es realizar una aproximación. Es decir, el sumatorio del genotipo del QTL por el efecto se cambia por el sumatorio del genotipo que se tiene del SNP por el efecto que se estima, asumiendo que se trata de una buena aproximación al valor genético del individuo. En realidad, si la situación fuera ideal, no se va muy desencaminado. Por lo tanto, el valor genético es el genotipo que tiene en cada uno de los SNPs en cada uno de los efectos que se estima en la población de referencia. Es la base de la selección genómica.
Hay una serie de métodos estadísticos, ya que calcular el valor genómico no es sencillo. Uno de los problemas más recurrentes es el de dimensionalidad. Normalmente se tienen más medidas de fenotipos que de genotipos, pero en esta ocasión es al revés, pero hay unos métodos estadísticos similares al BLUP con una compactación del número de marcadores para estimar los efectos.
Los beneficios de la selección genómica son que se acorta el intervalo generacional, ya que se puede tener una buena estimación al analizar el genotipado de un animal que puede ser desde su primer día de vida, se pueden utilizar las vías de mejora de machos y hembras y no sólo los machos, permite distinguir entre ‘hermanos completos’ y la fiabilidad es mayor que el índice de pedigrí.
CUESTIONES PRÁCTICAS
Hay una serie de cuestiones prácticas que son interesantes de entender en la selección genómica. También se pueden analizar como realidades o mitos de la genómica.
■ ¿Cuántos animales debería tener la población de referencia?
La población de referencia tiene que ser representativa de la población que queremos seleccionar, lo que implica que debe tener toda la variabilidad genética que existe en la población. Básicamente, si se estima efectos de segmentos cromosómicos, se deberían tener todo con una frecuencia más o menos proporcional a la que hay en la población de verdad. Por lo tanto, el tamaño de la población de referencia depende de si esa variabilidad es muy grande o no lo es. Cuanto mayor sea el tamaño efectivo de la población, mayor tendrá la población de referencia.
En principio, hay muchas fórmulas sobre la precisión, pero en una de ellas se conjuga el número total de registros de la población de referencia, la heredabilidad y la longitud del genoma. Realmente, la precisión depende del número de registros, por lo tanto del tamaño efectivo de la población de referencia. En función de que el tamaño efectivo sea de 100, 500 o 1.000, y la heredabilidad del carácter va desde 0,1 a 0,9. Si el carácter de producción de leche tiene una heredabilidad de 0,27 0 0,28 y el tamaño efectivo es de 100, necesitaríamos unos 15.000 animales, o si el tamaño efectivo es de 200, podríamos llegar a 40.000 animales de población de referencia.
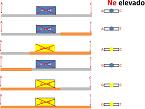
El tamaño efectivo depende de varias cuestiones. Hay que tener en cuenta que se parte de segmentos genómicos. Cuando se tiene un tamaño efectivo muy bajo, se tiene únicamente unos fragmentos cromosómicos. Si sólo se tienen dos fragmentos, únicamente se puede tener tres tipos de animales en la población combinando los resultados de esos dos fragmentos. Sin embargo, la realidad es más compleja. Además, los cromosomas intercambian material. Aquí interviene el tamaño de la población de referencia, ya que depende de los fragmentos que se tengan y su frecuencia. Si estuviera frecuente en la población, se tendría lo que se denomina ‘equilibrio de ligamiento’ y un tamaño efectivo grande, no se tendrían tres tipos de animales, sino 21 tipos. La variabilidad es muy grande. Con tamaño efectivo grande se da esa situación, con el agravante de que no se pueden distinguir esos 21 tipos, sino únicamente 9. La situación que suele darse es intermedia entre los tres tipos de animales o con poblaciones de tamaño efectivo grande.
Como conclusión, el tamaño ideal depende de la población. Se debe utilizar un número mayor de animales cuanto mayor sea el número de fragmentos cromosómicos o, como alternativa, utilizar un número más alto de marcadores para reducir la recombinación. Además de representar toda la variabilidad, también es positivo que haya un cierto parentesco entre los animales que se analizan en la población de referencia y los que se analizan para predecir.
■ La selección genómica funcionará igual en ovino de leche que en vacuno de leche.
En vacuno de leche, se aplicó la genómica en Holstein en Estados Unidos desde 2008. Sin embargo, Holstein tiene una situación perfecta, ya que es una población muy homogénea, con únicamente una raza que se utiliza para producir leche de vaca de forma global, con un uso masivo de la inseminación artificial. Además, la base de datos tiene un gran número de fenotipos, muy bien medidos y de manera uniforme, tiene un objetivo claro de selección y las organizaciones de criadores o las empresas de mejora son capaces de invertir en tecnología. Además, suele haber una evaluación genética centralizada y tiene un intervalo generacional largo, en ocho años a machos y algo más de dos en hembras.
En ovino de leche se ha comenzado a trabajar en Francia. Se tiene una situación muy lejana. En primer lugar, la población es muy heterogénea, con razas locales que tienen un tamaño efectivo normalmente más grande, en los casos de Lacaune, Sarda o Churra, muy por encima de Holstein en vacuno. La base de datos tiene un número de fenotipos bajo, los objetivos de selección no son uniformes en las diferentes razas, las organizaciones de criadores tienen unos recursos limitados, la evaluación genética está diversificada y el intervalo generacional es largo.
Si se observa el tamaño objetivo en vacuno y ovino, en el caso del ganado vacuno, tanto las razas abundantes oscilan entre 80 y 100, en ovino se habla de 300 o 400. La variabilidad que tenemos en ovino es bastante más grande que en vacuno.
En las tendencias de la selección genómica en Holstein en Estados Unidos, que tiene hasta un millón de hembras genotipadas, la tendencia es de una mejoría muy clara.
Por lo tanto, dadas las características de las razas de producción de leche de oveja, parece complicado llegar a las cifras de Holstein. Cada raza presenta una situación particular, por lo que dependerá de lo bien o mal que funcione la valoración genética clásica, y deberá plantear una población de referencia en función de la estructura de su población.
■ Es más fiable la valoración genómica que la tradicional.
Esta afirmación no es real. Si se coge el promedio de los padres cuando se realiza una valoración, la fiabilidad está en torno al 20% y el coste es muy bajo. Si se hace un test de descendencia, depende del número de corderos o hembras, oscilará entre 50% y 90% y el coste es alto. Por lo que respecta a la selección genómica, en el mejor de los casos se está en el límite inferior del sistema de valoración y se puede llegar hasta el 70%. El coste es bastante asumible. Se puede tener una buena fiabilidad al nacimiento, pero es tan buena como la valoración tradicional.
Sí es verdad que se pueden diferenciar los hermanos, que comparten un porcentaje de genoma entre el 40% y el 60%.
Es verdad que la selección genómica presenta fiabilidad intermedia entre el promedio de los padres y la valoración tradicional con un número limitado de hijas. Se obtiene a una edad temprana y permite acortar el intervalo generacional y la fiabilidad de la selección genómica dependerá de lo buena que sea la población de referencia y el esquema de selección.
■ Cuando se aplica selección genómica no hay que hacer medición de fenotipos.
No es una afirmación cierta. Se estima un efecto, pero las poblaciones evolucionan y el número de fragmentos cromosómicos no son iguales porque se van recombinando. Por lo tanto, se debe recalibrar de forma continua. En la tercera generación, se tiene una correlación del 85% con el verdadero valor genómico, pero en la octava generación ya es del 71%, por lo que se pierden un total de 14 puntos.
Así, las ecuaciones de estimación de los efectos de los segmentos cromosómicos deben ser actualizadas de forma permanente para reflejar la evolución de la población. Existen dos fuentes de información que hay que actualizar: relaciones familiares y estructura de la población.
Por lo tanto, la selección genómica no evita la medición de fenotipos y recalibrar las ecuaciones. Eso sí, puede permitir hacer la medición únicamente en la población de referencia.
■ La valoración inter-raza funcionará de forma aceptable.
Si un fragmento de 60 kilobases no es suficiente para detectar la variabilidad dentro de una raza, en las otras razas el número de segmentos es mucho mayor, por lo que no se va a tener una buena predicción si se aplica a varias razas. Se debería aumentar en gran medida la densidad de marcadores.
■ La selección genómica reduce el intervalo generacional.
La fórmula del progreso genético por año depende de la intensidad de la selección, de la precisión de los valores, de la desviación estándar y del intervalo generacional. Así, la selección tradicional en ganado vacuno coge el 5% mejor de los machos y la precisión es de 0,8 después de seis o siete años y no se mejora en hembras. Por lo tanto, cada año se progresa unos 0,2. En el caso de la genómica, se tiene menor precisión pero se gana en el intervalo generacional, por lo que se tendría el doble del progreso en cada año, que es algo que se va acumulando año tras año. Además, se mejora en los dos sexos.
PUNTOS FINALES
- La selección genómica funciona tanto mejor cuanto mejor sea el plan de mejora tradicional.
- La selección genómica no cambia ningún paradigma anterior.
- El uso de información genómica nos permite tener un programa más eficiente al acortar el intervalo generacional y al utilizar la presión de selección en ambos sexos.
Como reflexión general a cualquier tecnología, con el lanzamiento de una tecnología, se llega a un pico de expectativas sobredimensionadas. Poco después se cae en el ‘abismo de la desilusión’ al no cumplir esas expectativas tan amplias. Después se sube la ‘rampa de la consolidación’ para llegar a una ‘meseta de la productividad’.
Oviespaña, noticias diarias sobre el mercado nacional e internacional del ovino, investigación ganadera, alimentación y sistemas de manejo.